一图看懂单细胞文库Reads的组成成分
这篇文章是前两篇技术文档的后续:
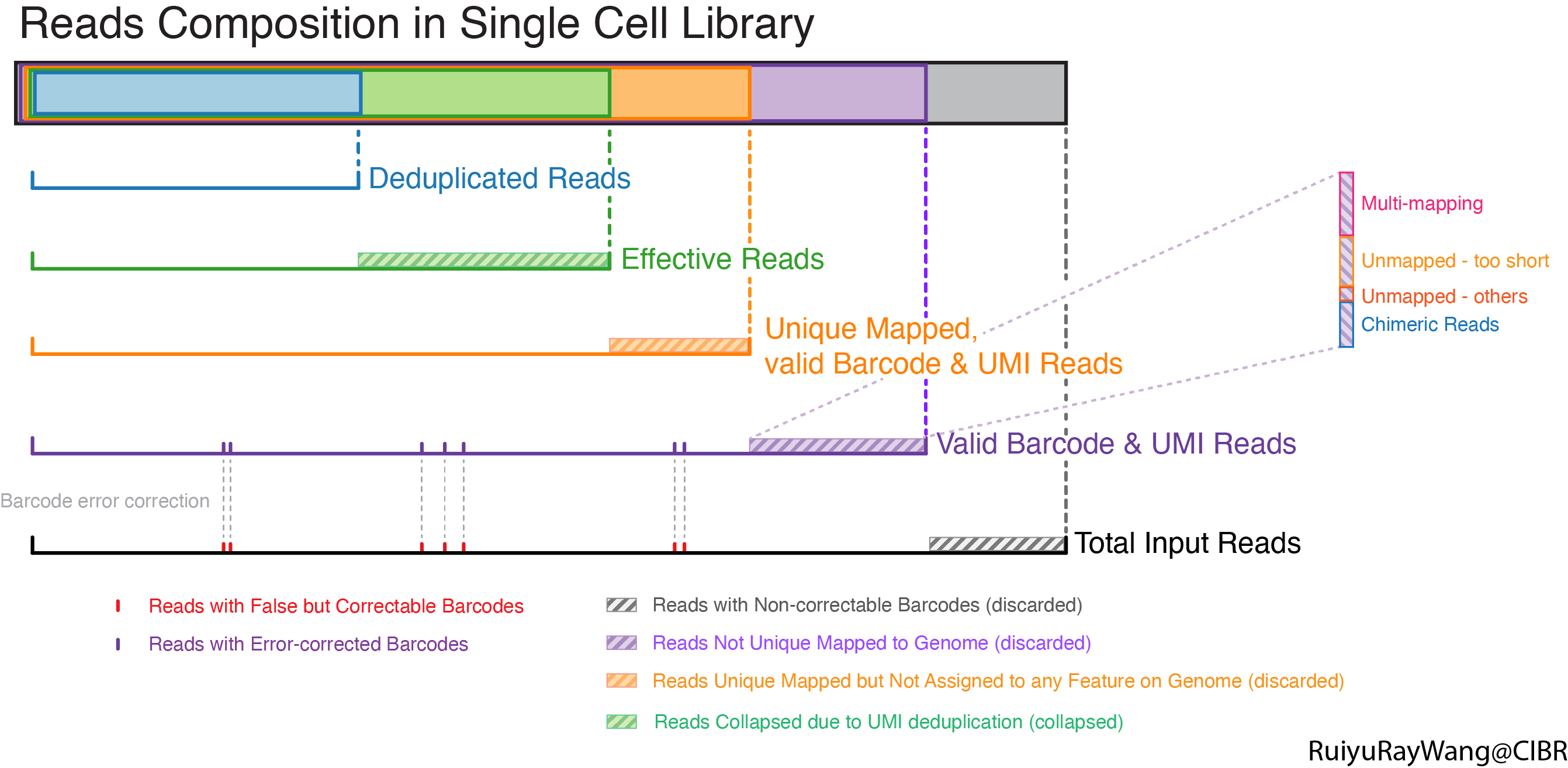
前两篇文章成文过程中我意识到,单细胞测序文库从Raw Data到Deduplicated Reads,其实是一个non-trivial的流程。从fastq文件下机开始,不同来源的Reads需经过层层计算和筛选,最终只有少部分Reads能保留在表达矩阵(或counts table)中。这里用一张图总结,以帮助我们更好地理解单细胞文库的建库和上游分析(Upstream Analysis)。
以ScRNAseq_smkpipe_at_Luolab的分析pipeline为例。
这套流程主要针对使用了Barcode + UMI的技术。
流程图读法:从下往上读,不同颜色代表不同分析阶段的Reads;画斜条纹阴影的线段表示当前步骤到下一步中被清除掉的Reads。
- 黑色:Total Input Reads,一般我用的是测序下机后的Clean Reads(公司给的QC质控后的Cleandata)
- 黑色->紫色:
umi_tools whitelist,wash_whitelist,umi_tools extract - 黑色阴影:Reads with Non-correctable Reads, invalid Barcode & UMI (discarded)
- 紫色:Barcode有效的Reads(Reads with valid Barcode & UMI)
- 紫色->黄色:
STAR - 紫色阴影:Reads NOT Unique Mapped to Genome (discarded)
- 黄色:Unique Mapped Reads,在10X中也叫Confidently Mapped Reads(Unique Mapped, valid Barcode & UMI Reads)
- 黄色->绿色:
featureCounts - 黄色阴影:Unique Mapped, valid Barcode & UMI but NOT assigned to feature
- 绿色:Effective Reads,即Unique Mapped, valid Barcode & UMI, feature assigned Reads
- 绿色->蓝色:
umi_tools count - 蓝色阴影:Reads Collapsed due to UMI deduplication
- 蓝色:Deduplicated Reads,即最终用于生成表达矩阵的对Reads计数的结果
有了这张图,我们可以做一个简单直观的推论:如果想提高单细胞测序的技术质量,则必须尽可能减少阴影部分的Reads在总文库中的比例。
仔细思考实验流程,会发现每个阴影部分会受不同因素的影响。
- 黑色阴影:主要影响因素之一是Barcode & UMI序列质量,即TSO引物质量;也有可能受TSO concatamer、RNA降解因素影响
- 紫色阴影:主要影响因素是样本制备质量,即细胞活率、异源RNA污染等
- 黄色阴影:主要影响因素是生物学样本内非mRNA成分的比例,如premRNA等,可能与样本本身生物学性质有关
- 蓝色阴影:主要与扩增循环数有关,该部分Reads过多可能是因为扩增循环数过高
“降低阴影部分比例”可以作为单细胞测序实验的一项指导原则,帮助我们进行实验条件的摸索,也可以作为单细胞技术开发的一项指导原则,帮助我们迭代优化试剂组成。
Last modified on 2022-02-20